נוקלאוזום
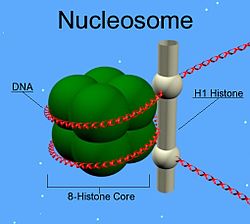
נוקלאוזום הוא תת-יחידה בסיסיות שחוזרת על עצמה פעמים רבות ברצף הכרומטין האיקריוטי (מלבד במקרה של תאי זרע). הנוקלאוזום אורז את ה-DNA לתוך כרומוזומים בתוך גרעין התא ושולט על בקרת הגנים.
הנוקלאוזום עשוי מ-DNA ומארבעה זוגות של חלבונים הנקראים היסטונים, והמבנה שלו מזכיר "חרוזים על חוט" כאשר צופים בו במיקרוסקופ אלקטרונים. ההיסטונים H2A, H2B, H3, ו-H4 הם חלק מהנוקלאוזום ואילו ההיסטון H1 מקשר בין שני נוקלאוזומים סמוכים.
השערת הנוקלאוזום, שהוצעה על ידי דון ועדה אולינס[1] ורוג'ר דויד קורנברג[2][3] ב-1974, הייתה דוגמה לשינוי שחל בהבנת הבקרה הגנית ביצורים איקריוטיים.
תפקוד הנוקלאוזום בגרעין
לנוקלאוזום יש שלושה תפקודים עיקריים בתוך גרעין התא:
- הוא מספק את רמת הדחיסות המזערית שדרושה כדי להחזיק את גדיל ה-DNA הכפול בתוך הגרעין.
- יש לו תפקיד בבקרת התעתוק ובמניעה מ-RNA פולימראז להיכנס לאזורי הקדם תעתוק של הגנים אם לתא אין צורך בתוצרי הגנים.
- אם דרישות התא משתנות, אנזימים מסוימים יכולים להזיז או לשנות את אופן הארגון של הנוקלאוזום כדי לאפשר גישה אל הגן. ישנה השערה כי לנוקלאוזום יש תפקיד ראשי בנשיאת מידע תורשתי אפיגנטי.
מבנה הליבה

המבנה הגבישי של הנוקלאוזום תואר ברזולוציה טובה יותר מ-2.0Å[4], אולם רוב המאפיינים הייחודיים שלו נודעו ב-1997 כאשר פורסם המבנה שלו ברזולוציה של 2.8Å[5].
הנוקלאוזום חוזר על עצמו, עם חריגות מסוימות, בערך כל 200 זוגות בסיסים לאורך הכרומטין האיקריוטי. חלקיק ליבת הנוקלאוזום שנראה באיור מורכב מבערך 146 זוגות בסיסים של DNA דו-גדילי שעטופים ב-1.65 סלילי על (supercoil) בכיוון שמאלה, מסביב לארבעה זוגות זהים של חלבונים בודדים הידועים כהיסטונים וביחד הם נקראים אוקטאמר ההיסטון. שאר 50 זוגות הבסיסים של היחידה החוזרת מורכבים מ-DNA מקשר, שהוא דו-גדיל המפריד בין שני חלקיקי ליבה.
לכל אחד מארבעת ההיסטונים (H2A, H2B, H3 ו-H4) יש מוטיב מבני דומה המורכב משלושה סלילי אלפא המופרדים על ידי לולאות. בתמיסה, ההיסטונים יוצרים זוגות עם עותקים זהים של עצמם ומתייחסים אליהם כאל דימרים או היסטונים מקופלים בזוגות. במקרה של ההיסטונים H3 ו-H4, הם ממשיכים להתאסף לטטראמרים, שהם חיבור של שני דימרים H3-H4, שבאמצעותם קבוצות טעונות מאותו סליל אלפא על שני ההיסטונים H3 נקשרים זה לזה בקשר מימן. ההתקבצות של חלקיק הליבה של הנוקלאוזום מתחילה כאשר הטטראמר H3-H4 נצמד ל-DNA הדו-גדילי ולאחר מכן נצמדים אליו שני דימרים נפרדים מסוג H2A-H2B, תהליך שסביר להניח שיתרחש באופן שיתופי (כלומר, שני הדימרים H2A-H2B מתקבצים לטטראמר בבת אחת).
לפי המבנה הגבישי שלו, ההיסטון האוקטאמרי קשור ל-DNA שמסביבו בערך כל 10 זוגות בסיסים. כל אחד מארבעת הדימרים של ההיסטון מכיל שלושה אזורי קשר עם ה-DNA. אתר הקישור המרכזי בכל דימר נוצר על ידי סליל אלפא מכל היסטון שבצמד המצביע לכיוון קבוצת זרחה בודדת ב-DNA שאליו הם קשורים בקשר מימן. בעמדות של 10 זוגות בסיסים לכל כיוון, לולאה משני ההיסטונים מתלכדת לקשר מימן לקבוצות זרחה אחרות. שני קשרים אחרים (מתוך סך של 14) נוצרים בעקבות הקישור של זנבות ההיסטונים מסוג H3. קשרים אלה קיימים בנקודות הכניסה והיציאה של ה-DNA שעטוף סביב הנוקלאוזום ומסייעים בהידוק האזורים האלה אל חלקיק הליבה.
ניתוח של מבנה גדיל ה-DNA הכפול העטוף מסביב לאוקטאמר ההיסטוני מראה שצורתו היא בדרך כלל מסוג B, אף על פי שהמבנה יותר מוגבל מאשר DNA חופשי בגלל הקשרים עם האוקטאמר. ההתעקמות לתוך סליל העל נובעת בעיקר מכך שהחריץ העיקרי מופיע מול האוקטאמר ולפיכך היא מתרחשת כל חמישה זוגות בסיסים בקירוב. החריץ העיקרי שמקיף את האוקטאמר הוא חלק. חריצים קטנים יותר מחוזקים על ידי שרשראות צד של ארגינין שחודרות לתוך החריץ ומופיעים בצורה חלקה סביב הטטראמר H3-H4, אבל מתכופפים סביב אזורי הדימר H2A/H2B. ה-DNA מצוי בצורתו ההדוקה ביותר באזורים שבהם הוא קשור עם מבני הלולאה הכפולה של דימרי ההיסטון שהוזכרו לעיל, מה שמרמז על כך שיש יותר גיוון בצורה שבה ה-DNA קשור למבני הסליל אלפא של דימרי ההיסטון על מנת להשיג את הקשירה של רצפים שונים[6].
חלבונים רבים נקשרים לרצפי DNA ייחודיים בלבד. אף שהנוקלאוזומים נוטים להעדיף מספר רצפי DNA על פני אחרים, הם יכולים להיווצר על כל רצף שהוא. מספר ניסויים הראו שמולקולות מים מכפילות פי שניים בקירוב את מספר קשרי ההיסטון-DNA באמצעות מילוי תפקיד של מתווכות בין אטומים שבמקרה אחר רחוקים זה מזה מכדי ליצור קשר מימן[4]. גמישות זו ביצירת קשרים מתווכי מים מאפשרת לאוקטאמר ההיסטוני לעטוף מגוון רחב מאד של רצפי DNA.
המבנה והיעוד של זנבות ההיסטון
הקצה של כל היסטון מכיל זנב של שיירי חומצות אמינו באורכים שונים, האופייניים לכל היסטון. מטרת הזנבות איננה ברורה לחלוטין כיום, אולם נראה שהם תורמים ליציבות הנוקלאוזום[7] ומשמשים גם כאתרי עגינה עבור חלבונים אחרים. מבנה הזנבות עשוי להשתנות קלות על ידי אנזימים אחרים בגרעין ועשוי למלא תפקיד משמעותי ביצירת מבני כרומטין מסדר גבוה[8].
מבנה מסדר גבוה

בגרעין התא יש צורך בדחיסה נוספת של הכרומטין, אולם היא לא מובנת לחלוטין. צורת ההבנה הנוכחית[9] היא שרצף נוקלאוזומים עם קטעי DNA "מקשרים" יוצר סיב בגודל 10nm, המכונה "חרוזים על חבל", שיש לו יחס אריזה של ~6, בהשוואה ל-DNA חופשי (לכל ננומטר אורך). שרשרת של נוקלאוזומים יכולה להתארגן בסיב 30nm, מבנה יעיל (נחשב כבעל צורה של סולנואיד סלילי, מבנה סרט מזוגזג, חרוז-על, או חסר מבנה) עם יחס אריזה של ~40.
במספר ניסויים הצליחו להפיק גביש שהמבנה שלו הוא דמוי טטרא-נוקלאוזום ונעשה בו שימוש כדי לבנות סיב של 30nm[10]. התוצאה מניבה עדות חזקה שתומכת בדגם של סליל דו-התחלתי, שבו הנוקלאוזומים מאורגנים בסרט זיגזגי שמתפתל לסלילי על. ההיסטון H1 מייצב את סיב ה-30nm.
מעבר לרמה זו, מבנה הכרומטין מאד לא מובן, אולם הוצע שסיב ה-30nm מתארגן לתוך לולאות לאורך חלבון פיגום מרכזי, וכתוצאה מכך נוצר אאוכרומטין שניתן לתעתוק. דחיסה נוספת תגרום ליצירת הטרוכרומטין, שאותו לא ניתן לתעתק.
עיצוב מחדש של הנוקלאוזום
מספר תצפיות הראו שישנם אנזימים מסוימים המשנים את עמדת הנוקלאוזומים בניסוי (in vitro)[11]. היעוד שלהם הוא לחשוף מידע גנטי שמוחזק בתוך חלקיק הליבה של הנוקלאוזום כאשר התא זקוק לו. הוצע כי הנוקלאוזומים המעוצבים מחדש משנים לא רק עמדות על תבנית ה-DNA אלא שיש להם מבנים אחרים יציבים או יציבים למחצה. המצבים המשתנים הללו עשויים להיות נחוצים כדי שהתעתוק יתאפשר.
מוטציות תלויות Swi/Snf
יצירה של SWI/SNF, שהם אנזימי הארגון מחדש של הנוקלאוזום, חיונית להישרדותם של שמרים[12]. אולם, ניתן להתגבר על מגבלה זו באמצעות מוטציות בשייר אחד של ההיסטון H3 או של ההיסטון H4. המבנה הגבישי של 11 מוטציות כאלה תואר[13] וייתכן שהוא ייתן מידע על האופן שבו SWI/SNF מספקים גישה לרצפים גנטיים שלא הייתה אליהם גישה בגלל שהיו עטופים סביב הנוקלאוזום.
התקבצות הנוקלאוזום בניסוי

נוקלאוזומים יכולים להתקבץ בניסוי באמצעות שימוש בהיסטונים טבעיים מזוקקים או היסטונים רקומביננטיים או במגוון המבנים השונים שלהם[14]. השיטה התקנית של שימוש בדיאליזת מלח נשענת על העובדה שהטטראמר H3-H4 נקשר ל-DNA בריכוזי מלח גבוהים יותר מאשר הדימר H2A/H2B.
תגובה המורכבת מארבע היסטוני ליבה ותבנית DNA עירומה מבוצעת בהתחלה בטמפרטורה של 4C בריכוז מלח של 2 מולאר. הריכוז הזה יורד בהדרגתיות באמצעות החלפה ידנית של בופר, או באמצעות הזרמה של בופר בריכוז מלח נמוך לתוך התגובה כאשר במקביל שואבים החוצה את תערובת התגובה באותו קצב. ככל שריכוז המלח יורד, טטראמרים חופשיים בתמיסה מתחילים להיקשר עם תבנית ה-DNA. כאשר ריכוז המלח יורד עוד, הדימרים H2A/H2B נקשרים עם הטטראמר שעל התבנית.
קישורים חיצוניים
הערות שוליים
- ↑ Olins AL and Olins DE, "Spheroid Chromatin Units (nu Bodies)", Science (1974); 183: 330 - 332
- ↑ McDonald D, "Milestone 9, (1973-1974) The nucleosome hypothesis: An alternative string theory", Nature Milestones: Gene Expression. (2005) Dec 1; [1]
- ↑ Kornberg, RD, "Chromatin structure: a repeating unit of histones and DNA", Science. (1974); 184: 868–871
- ^ 4.0 4.1 Davey CA, Sargent DF, Luger K, Maeder AW, Richmond TJ, "Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 Å resolution.", Journal of Molecular Biology. 2002 Jun 21; 319 (5): 1097-1113.
- ↑ Luger K, Mader AW, Richmond RK, Sargent DF, Richmond TJ, "Crystal Structure of the Nucleosome Core Particle at 2.8 Å Resolution", Nature. 1997 Sep 18; 389 (6648): 251-60.
- ↑ Richmond TJ, Davey CA, "The structure of DNA in the nucleosome core", Nature. (2003) May 8; 423 (6936): 145-150.
- ↑ Brower-Toland B, Wacker DA, Fulbright RM, Lis JT, Kraus WL, Wang MD, "Specific contributions of histone tails and their acetylation to the mechanical stability of nucleosomes", Journal of Molecular Biology (2005) Feb 11; 346 (1): 135-146
- ↑ Luger K, Richmond TJ, "The histone tails of the nucleosome.", Current Opinion in Genetics and Development. (1998) Apr; 8 (2): 140-6.
- ↑ Chakravarthy S, Park YJ, Chodaparambil J, Edayathumangalam RS, Luger K, "Structure and dynamic properties of nucleosome core particles", FEBS Letters. (2005) Feb 7; 579 (4): 895-898.
- ↑ Schalch T, Duda S, Sargent DF, Richmond TJ, "X-ray structure of a tetranucleosome and its implications for the chromatin fibre", Nature. (2005) Jul 7; 436: 138-141.
- ↑ Lia G, Praly E, Ferreira H, Stockdale C, Tse-Dinh YC, Dunlap D, Croquette V, Bensimon D, Owen-Hughes T, "Direct Observation of DNA Distortion by the RSC Complex", Molecular Cell (2006) Feb 3; 21 (3): 417-425.
- ↑ Kruger W, Peterson CL, Sil A, Coburn C, Arents G, Moudrianakis EN, Herkowitz I, "Amino acid substitutions in the structured domains of histones H3 and H4 partially relieve the requirement of the yeast SWI/SNF complex for transcription, Genes Development. (1995) Nov 15; 9 (22): 2770-2779.
- ↑ Muthurajan UM, Bao Y, Forsberg LF, Edayathumangalam RS, Dyer PN, White CL, Luger K, "Crystal structures of histone Sin mutant nucleosomes reveal altered protein-DNA interactions", EMBO Journal (2004) Jan 28; 23 (2): 260-271.
- ↑ Dyer PN, Edayathumangalam RS, White CL, Bao Y, Chakravarthy S, Muthurajan UM, Luger K, "Reconstitution of nucleosome core particles from recombinant histones and DNA", Methods in Enzymology (2004); 375: 23-44