סמיילס
Simplified Molecular-Input Line-Entry System ובראשי תיבות SMILES היא שיטת סימול ליניארית ופשוטה לייצוג מולקולות.
SMILES היא שיטת סימול לייצוג של צורונים כימיים באופן ממוחשב עבור צרכים של עיבוד מידע, תכנות ומידול בתחום הכימיה.[1] השיטה מבוססת על עקרונות התיאור הגרפי הדו-ממדי של מולקולות על ידי שימוש באותיות ובסמלים כך שיתארו את הצמתים ואת הקצוות של הגרף, קרי, את האטומים ואת הקשרים הכימיים.[2] הסמלים, מבוססי ASCII, מסודרים לאורך קו (line notation) בסדר מסוים, כלומר, מחרוזת של סמלים ואותיות (characters) המתארת בצורה חד משמעית את המבנה הטופולוגי של המולקולה.
SMILES נחשבת לשיטה אינטואיטיבית ופשוטה לקריאה המובנת גם למחשב וגם למשתמש שאינו כימאי בהכרח.[1] כדי לוודא שהמחשב יוכל להבין באיזה צורון מדובר, ישנם כללי כתיבה ברורים למחרוזת SMILES שיש להקפיד עליהם על מנת שהמולקולה תתואר בצורה נכונה. סימול SMILES משמש בין היתר לייצוג של מולקולות במאגרי מידע שונים ולחיפושן באמצעות אלגוריתמים. בנוסף, נעשה שימוש בSMILES במגוון תחומי כימיה כגון מידול מנגנונים קינטיים בתהליכים כימיים ולמידת מכונה (machine learning) לצורכי חישובים וניבויים של מערכות מולקולריות.[1][3]
היסטוריה
מפרט SMILES המקורי נכנס לשימוש בשנות ה-80, ומאז הוא שונה והורחב. היזם של שיטת SMILES היה דייוויד ווינינגר (אנ') שפעל בשיתוף עם הסוכנות האמריקנית להגנת הסביבה.[4][5] כימאים וחוקרים נוספים תרמו גם הם לפיתוח הראשוני של SMILES וכן לעדכונים ולשינויים שנערכו בשיטה במרוצת השנים, שרבים מהם בוצעו על ידי חברתDaylight Chemical Information Systems.[6] בשנת 2007 פותח תקן פתוח בשם OpenSMILES בקהילת הכימיה Blue Obelisk שעושה שימוש בקוד פתוח.
ישנן שיטות ליניאריות נוספות כגון Wiswesser Line Notation (WLN) ואחרות. שיטה פופולרית נוספת לקידוד מולקולות בצורה קווית היא שיטת InChI שפותחה ב-2006 על ידי IUPAC, אך נחשבת לפחות אינטואיטיבית לקריאה עבור המשתמש.
תכונות
SMILES יכול לתאר מחרוזת בודדת או מערך של מחרוזות המתארות מספר מולקולות. בנוסף, מחרוזות שונות יכולות לתאר את אותה המולקולה: אתנול, למשל, יכול להיות מתואר כ CCO, OCC ו-C(O)C. כלומר, SMILES אינה שיטה קנונית, מה שיכול להוות בעיה בקידוד ולהאריך את זמן החיפוש בהינתן מאגר נתונים גדול.
יתר על כן, חוסר הקנוניות מקשה על התוכנה בחיפוש ובקידוד של מבנים שונים ושל תת-מבנים מולקולריים. כדי להתגבר על הבעיות הללו, ישנם אלגוריתמים המפיקים SMILES קנונים כמו OpenEye Scientific Software ו-Universal Smiles. כלומר, בעיית הקנוניות היא תלוית-אלגוריתם. בנוסף, SMILES יכולה לתאר את הסידור המרחבי של המולקולה במבנה של טטראדר או של קשר כפול וכן של איזומרים שונים ואף לאפיין כיראליות של מולקולות, כפי שמפורט בכללים מטה.
כללי הכתיבה של SMILES
אטומים נכתבים ב-SMILES באמצעות אותיות גדולות, ואליהן מתלוות אותיות קטנות, אם היסוד הכימי מכיל יותר מאות אחת. החוק הבסיסי של SMILES הוא הכללת אטומי המימן באופן אוטומטי, כך שאין צורך לציינם באופן מפורש, על אף שבפועל ניתן להוסיף אותם. ישנם מקרים מיוחדים שבהם יש לציינם באופן מפורש, לדוגמה רדיקלים כימיים.
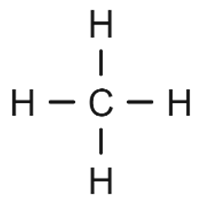
כדוגמה לסימול מולקולה פשוטה, מולקולת מתאן תסומן כ-C בלבד. סימול זה מציין פחמן עם ארבעה קשרים יחידים קוולנטיים לארבעה מימנים (דוגמה 1 בטבלה מטה). מנגד, אם מדובר בצורון שהוא יסוד כימי או רדיקל, או שאינו האיזוטופ השכיח, יש להוסיף סוגריים מרובעים לצורון אם מידע נוסף רלוונטי. להלן, אטום פחמן יסומן על ידי [C] (דוגמה 2).
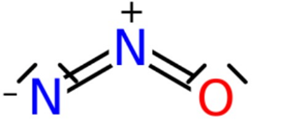
דוגמה נוספת היא ייצוג של קטיון טיטניום Ti4+ בשני אופנים זהים: [Ti+4] או לחלופין [Ti++++] (דוגמה 3). ניתן אף לייצג מטענים פורמליים במולקולה, כגון N2O כפי שמופיע בדוגמה 4. דוגמה למולקולה עם רדיקל היא אתנול עם רדיקל על הפחמן הנמצא בקצה המולקולה (דוגמה 5).[7]
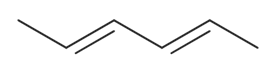
- קשרים קוולנטיים בין שני אטומים מהסוגים: יחידים, כפולים ומשולשים, מתורגמים ב-SMILES לסימנים -, =, # ,בהתאמה. קשר קוולנטי בודד יכול להיות מיוצג ללא מקף כלל ואכן הנוהג הוא לא לציינו. לדוגמה, מולקולת אתאן המיוצגת כך: CC, זהה מבחינה סמנטית לC-C (דוגמה 6). דוגמה נוספת למולקולה עם קשרים יחידים וכפולים לסירוגין היא הקסדיאן (2,4-hexadiene) (דוגמה 7).
- קשרים נוספים הניתנים לייצוג על ידי SMILES, הם קשרים יוניים וקשרי ואן-דר-ואלס המסומנים בנקודה (דוגמה 8 עבור נתרן כלורי). ניתן אף לייצג קשרים מרובעים על ידי הסימן $.
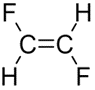
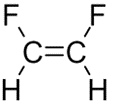
- סטריאוכימיה מיוצגת על ידי לוכסן ימני (/) או שמאלי (\) לקשר קוולנטי יחיד מסוג טראנס (trans) או ציס (cis) הסמוך לקשר כפול. שני לוכסנים עוקבים בהטייה זהה, מייצגים קשר טראנס ואילו שני לוכסנים בהטיות מנוגדות מייצגים קשר ציס (דוגמאות 9 ו-10).
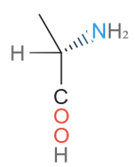
- מבחינת סטריאוכימיה כיראלית, ישנה דרך לייצג מולקולות במבנה טטראדרלי זהה, הנקראות אננטיומרים, אך מהוות תמונת ראי אחת של השנייה. ייצוג של סיבוב המרחב עם כיוון השעון מסומן על ידי הסימון @@ ואילו סיבוב נגד כיוון השעון מסומן על ידי הסימן @. לדוגמה, מולקולת אלאנין בשתי הקונפיגורציות בדוגמאות 11 ו-12 בטבלה.
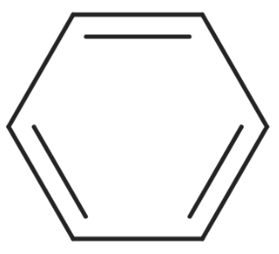
- על מנת לייצג טבעת כימית (טבעת בעלת שלושה אטומים לפחות) בתצורת SMILES, יש לפצל את הטבעת במיקום שרירותי על מנת לסמן את ההתחלה ואת הסוף של הטבעת באמצעות תווית נומרית. לדוגמה, מולקולת בנזן תסומן על ידי C1=CC=CC=C1 (דוגמה 13). התוויות הספרה "1" משמעה ששני האטומים הסמוכים משמאל לתוויות מחוברים גם הם. אם מדובר בטבעות בי-ציקליות, ניתן לפצל את הטבעת הראשונה במיקום שרירותי עם אינדקס 1, ואילו את הטבעת השנייה לפצל עם אינדקס 2. להלן, מולקולת דקאלין תיכתב ב-SMILES באופן הבא: C1CCCC2C1CCCC2. צורה נוספת לרישום מולקולה זאת, היא C1CCCC2CCCCC12. בסימול זה, הפחמן האחרון מייצג סגירה של טבעות 1 ו-2, כל אחת לחוד (דוגמה 14). אם מדובר במולקולה בעלת מספר טבעות העולה על 9, תחילת האינדקס יסומן על ידי הסימן % לפני המספור עצמו. לדוגמה, הסימון: %10 מציין כי מתייחסים לטבעת מספר 10 במולקולה, ולא להתחלה או לסוף של טבעות 0 ו-1. מומלץ להשתמש באינדקסים שהם מספרים עולים ועוקבים על מנת שלמשתמש יהיה קל ונוח יותר לקרוא את ייצוג ה-SMILES של המולקולה.
- ארומטיות ניתנת לייצוג על ידי אחת משתי הדרכים הבאות:
- ייצוג קשרים יחידים וכפולים לסירוגין, להלן, מקרה של מולקולת בנזן C1=CC=CC=C1 (דוגמה 13) בהתאם לשיטת מבני קקולה.
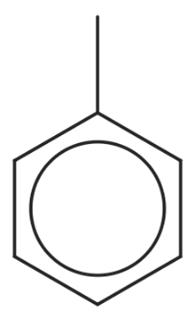
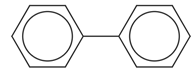
- ייצוג הטבעת הארומטית על ידי אותיות קטנות, להלן, המקרה של מולקולת טולואן, המיוצג בתור Cc1ccccc1 (ששת הפחמנים של הטבעת מיוצגים באותיות קטנות ואילו המתמיר המתילי שלה מיוצג בפחמן באות גדולה, דוגמה 15). ייצוג זה מתאים לשיטת קלאר עבור מולקולות פוליציקליות ארומטיות.[8] במולקולות ארומטיות ממשפחת פירול, במקרה בו ישנו קשר קוולנטי יחיד בין חנקן למימן במולקולה ארומטית, יש לציין זאת באופן הבא: [nH]. דוגמה למולקולה כזאת היא אימידאזול שהייצוג שלה יהיה n1c[nH]cc1 (דוגמה 16). במקרה בו טבעות ארומטיות מחוברות בקשר יחיד ביניהן, כגון ביפניל, יש לציין קשר יחיד זה במפורש ב-SMILES. עבור ביפניל, הייצוג יהיה: c1ccccc1-c2ccccc2 (דוגמה 17).
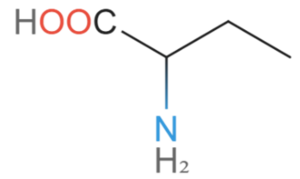
- אטומים מותמרים על טבעת או על שלד המולקולה יסומנו בתוך סוגריים עגולים. לדוגמה, מולקולת איזובוטאן תיוצג על ידי CC(C)C (דוגמה 18). דוגמה נוספת לסיעוף היא הוספת שייר קרבוקסילי ואמיני לשלד פחמימני במולקולה אלאנין. מולקולה זאת תירשם בצורה הבאה: CCC(N)C(=O), כאשר השייר האמיני מסומל על ידי (N) והשייר הקרבוקסילי מסומל על ידי (O=). השלמת המימנים מתבצעת באופן אוטומטי (דוגמה 19).
- מתמירים על גבי טבעת יכולים להיכתב באופן הנזכר לעיל, במיקום המתאים הנמצא בתוך סימון הטבעת. לחלופין אפשר לא לכתוב את המתמיר של הטבעת בסוגריים עגולים אם המתמיר נכתב לפני פתיחה או סגירה של הטבעת. לדוגמה מולקולת טולואן (דוגמה 15).
- על מנת לייצג איזוטופ מסוים, יש לכתוב בסוגריים מרובעים את מספר האיזוטופ לפני סימול היסוד. לדוגמה, פחמן 14 ייצוג על ידי [14C]. בנזן עם פחמן יחיד מסוג פחמן 14, יסומן באופן הבא: [14c]1ccccc1.
דוגמאות למולקולות שונות וייצוגן ב-SMILES[9]
הרחבות של SMILES
ישנן מספר הרחבות מבוססות SMILES אשר נועדו לדייק ולהקל על השימוש. אחת מהן היא SMARTS, שיטה המאפשרת חיפוש של תת-מבנים ושל מבנים מולקולריים במאגרי נתונים באמצעות מחרוזת SMILES מסוימת. למשל, כדי לחפש מבנים של מולקולות המכילות פנול, ניתן להשתמש במחרוזת SMARTS הבאה: [OH]c1cccccc1. השיטה כוללת אופרטורים לוגיים ומתארי מולקולות בנוסף לאלו של SMILES.[1][10]
הרחבה נוספת היא SMIRKS שמתארת תגובות גנריות, כלומר קבוצה של תגובות שקורה בהן תהליך זהה. תגובות מתוארות על ידי הפרדה בין מגיבים, תוצרים, ומחוללי תגובה באמצעות הסימן <, גם ב-SMIRKS וגם ב-SMARTS. ניתן להשאיר שדות ריקים בתגובה. במקרה שבו רוצים לכלול מספר מולקולות, מפרידים באמצעות נקודה. SMIRKS היא למעשה הכלאה של SMILES ו-SMARTS שנועדה לענות על הצורך הכפול של תגובה גנרית: ביטוי התגובה וביטוי של השפעות עקיפות לתגובה, כגון אטומים שלא משתתפים באופן ישיר בתגובה.[11][12]
ראו גם
קישורים חיצוניים
 אתר האינטרנט הרשמי של סמיילס
אתר האינטרנט הרשמי של סמיילס- חיפוש מולקולות באמצעות SMILES, באתר rmg
- מאגר לחיפוש צורונים כימיים, באתר PubChem
- תרגיל כתיבת SMILES, באתר EPA
הערות שוליים
- ^ 1.0 1.1 1.2 1.3 Wigh DS, Goodman JM, Lapkin AA. A review of molecular representation in the age of machine learning. Wiley Interdiscip Rev Comput Mol Sci. 2022;12(5):1-19. doi:10.1002/wcms.1603
- ^ Daylight Theory: SMILES, www.daylight.com
- ^ Quirós M, Gražulis S, Girdzijauskaitė S, Merkys A, Vaitkus A. Using SMILES strings for the description of chemical connectivity in the Crystallography Open Database. J Cheminform. 2018;10(1):1-17. doi:10.1186/s13321-018-0279-6
- ^ David Weininger, SMILES, a chemical language and information system. 1. Introduction to methodology and encoding rules, Journal of Chemical Information and Computer Sciences 28, 1988-02-01, עמ' 31–36 doi: 10.1021/ci00057a005
- ^ Office of Research & Development, SMILES (SIMPLIFIED MOLECULAR IDENTIFICATION AND LINE ENTRY SYSTEM): A LINE NOTATION AND COMPUTERIZED INTERPRETER FOR CHEMICAL STRUCTURES, cfpub.epa.gov (באנגלית)
- ^ Daylight Theory: SMILES, www.daylight.com
- ^ David Weininger, SMILES, a chemical language and information system. 1. Introduction to methodology and encoding rules, Journal of Chemical Information and Computer Sciences 28, 1988-02-01, עמ' 31–36 doi: 10.1021/ci00057a005
- ^ Hansen P, Zheng M. The Clar number of a benzenoid hydrocarbon and linear programming. J Math Chem. 1994;15(1):93-107. doi:10.1007/BF01277551
- ^ Probst D, Reymond JL. SmilesDrawer: Parsing and Drawing SMILES-Encoded Molecular Structures Using Client-Side JavaScript. J Chem Inf Model. 2018;58(1):1-7. doi:10.1021/acs.jcim.7b00425
- ^ Daylight Theory: SMARTS - A Language for Describing Molecular Patterns, www.daylight.com
- ^ Daylight Theory: SMIRKS - A Reaction Transform Language, www.daylight.com
- ^ 5.8: Line Notation (SMILES and InChI), Chemistry LibreTexts, 2020-08-11 (באנגלית)
36466126סמיילס